RNAi und CRISPR: Leistungsvergleich und Anwendungsleitfaden
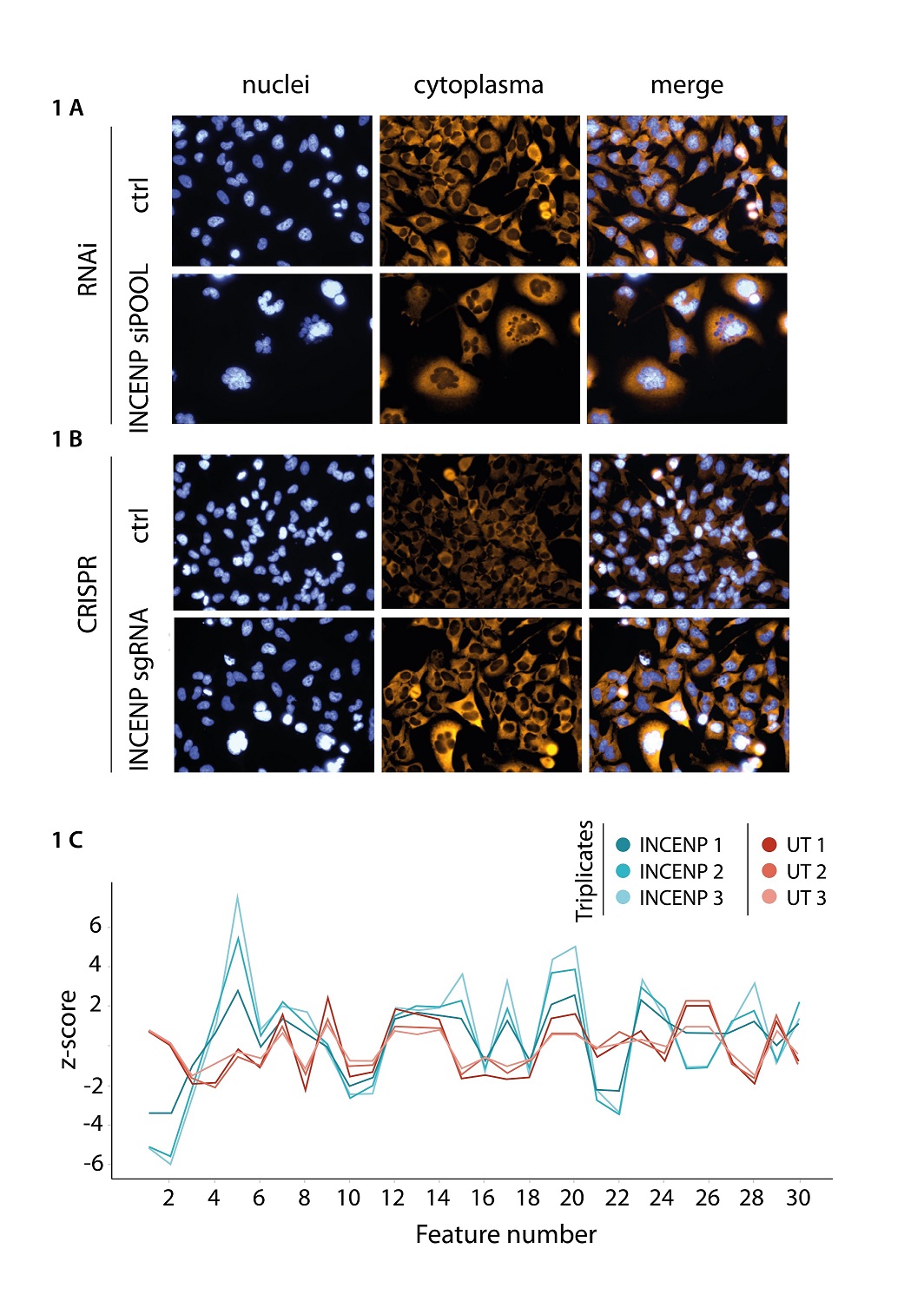
Abb. 1A: fluoreszenzmikroskopische Aufnahmen von Zellen in denen das Gen INCENP mittels RNAi dereguliert wurde. Die obere Bildreihe zeigt unbehandelte Kontrollzellen (Zellkerne blau, Zytoplasma orange). Die behandelten Zellen und Zellkerne (untere Bildreihe) zeigt Zellen, die in Anzahl, Größe und Morphologie signifikant verändert sind Abb. 1B: fluoreszenzmikroskopische Aufnahmen von Zellen in denen das Gen INCENP mittels CRISPR dereguliert wurde. Gegenüber den Kontrollzellen in der oberen Bildreihe sind nur wenige Zellen und Zellkerne bezüglich Größe und Morphologie verändert. Die überwiegende Anzahl der Zellen wurden von CRISPR nicht editiert Abb. 1C: Multiparametrische automatisierte Bildanalyse von Zellen in denen INCENP mit siPOOLs dereguliert wurde. 30 Parameter generieren für unbehandelte Zellen (rote Graphen) und behandelte Zellen (blaue Graphen) signifikante Profile. (Quelle: siTOOLs Biotech GmbH)
Mit RNAi und CRISPR stehen zwei unterschiedliche Methoden zur Gen-Funktionsanalyse zur Verfügung. Beide Methoden können die Genexpression unterdrücken, wobei RNAi auf Ebene der mRNA wirkt und beim CRISPR-Verfahren die DNA editiert wird. Technisch unterscheiden sich die Ansätze grundsätzlich und liefern oft widersprüchliche Ergebnisse. Ein Ziel des Projekts ist, mit einer vergleichenden Leistungsprüfung die Grenzen der beiden Methoden aufzuzeigen, sinnvolle Kombinationen vorzuschlagen und Nutzern einen Leitfaden zum sinnvollen Einsatz der Verfahren zur Verfügung zu stellen.
In einer Zusammenarbeit zwischen dem Münchner Biotech Unternehmen SiTOOLs Biotech GmbH und Prof Dr. H. Leonhardt (LMU München) wurden siRNAs und CRISPR-Reagenzien an einer Auswahl von Genen systematisch getestet und die Ergebnisse bioinformatisch ausgewertet.
Dazu wurden kultivierte Zellen nach Behandlung mit den Testreagenzien auf einem automatisierten Fluoreszenzmikroskop untersucht. Auf Basis einer multiplexen Analyse mit parallel ausgelesenen Fluoreszenzfarbstoffen konnte durch bioinformatische Auswertung das Ansprechverhalten der Zellen auf die Reagenzien als spezifische Signatur miteinander verglichen werden.
Während siRNAs mit hoher Effizienz beinahe jede Zelle treffen, wird mit CRISPR eine sehr niedrige Penetranz erreicht. Da in gemischten Zellpopulationen spezifische zelluläre Antworten nicht nachweisbar sind, müssen monoklonale Zellen erzeugt werden, die erst nach aufwendiger Selektion und Expansion funktionell untersucht werden können.
Zusammenfassend sind einzeln eingesetzte siRNAs nicht in der Lage, verlässlich spezifische Gene zu deregulieren, um auf deren Funktion zu schließen. Durch die Nutzung komplexer siRNA-Mischungen (siPOOLs) werden off-target-Effekte signifikant reduziert, aber nicht vollständig ausgeschlossen. CRISPR bedarf aufgrund von Adaptation zusätzlicher Kontrollexperimente.


 Leichte Sprache
Leichte Sprache